清华新闻网1月13日电 冷冻电子断层成像(cryo-electron tomography,cryoET)技术、冷冻聚焦离子束(cryo-focused ion beam,cryoFIB)样品减薄技术和子断层平均技术(sub-tomogram averaging,STA)的进步推动了生物大分子在细胞中的原位结构测定,对于研究膜蛋白结构及其在细胞环境中的相互作用具有重要意义。断层扫描图像(tomogram)是三维图像,但由于低信噪比和细胞内拥挤的环境,当前的观察方法仍主要依赖查看tomogram中的二维截面。然而,细胞中的膜结构通常呈弯曲形态,这使得通过二维截面难以直接观察膜表面蛋白的分布。同时,膜信号强烈的衬度也会干扰膜蛋白的识别,增加原位观察和结构解析的难度。
1月8日,清华大学生命学院李雪明副教授课题组在《自然·通讯》(Nature Communications)在线发表题为“MPicker:冷冻电子断层成像中膜蛋白的可视化与挑选”(MPicker:visualizing and picking membrane proteins for cryo-electron tomography)的研究论文。该论文报道了一种基于“膜展平”的方法,通过在“展平tomogram”中进行操作来降低膜蛋白可视化与挑选的难度(图1)。该方法首先通过膜分割或者手动标注定位感兴趣区域(一片膜),然后在tomogram中对这片膜及其周围的区域进行变形处理,最终把这个局部区域展平为一个包含平整膜结构的新tomogram,也就是展平tomogram。在这个新生成的展平tomogram中,原本弯曲的膜会变成一个平板,膜厚度与蛋白取向局部保持不变。展平tomogram中每一层二维截面都对应一个平行于膜的等距面,使得膜表面的蛋白信号可以轻易地以二维图像的形式被展示。这起到了降维的效果,降低了膜蛋白分析的空间复杂性,改善了膜上和膜周围蛋白质的可视化效果。
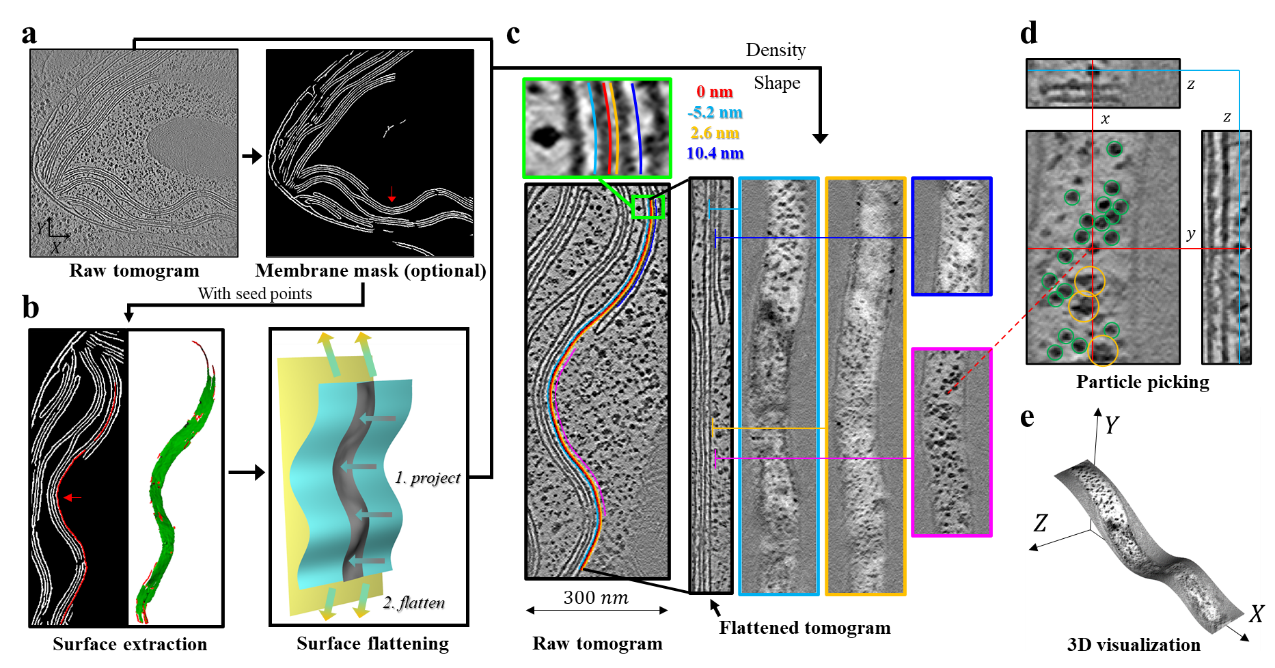
图1.MPicker软件流程图
基于膜展平方法,研究团队开发了名为MPicker的新软件,该软件能够在多种细胞切片样品的tomogram中清晰展示膜表面的蛋白分布(图2)。在展平tomogram中,用户可以更方便地识别并挑选颗粒。为了提高自动化程度,MPicker还整合了EPicker软件(一款原本用于单颗粒图像的自动颗粒挑选工具),充分利用了展平tomogram二维化的优势。除了在MPicker中进行操作,用户也可以灵活地使用其他软件在展平tomogram中进行颗粒挑选。

图2.使用MPicker对类囊体膜进行展平
MPicker还可以提供每个颗粒的初始取向用于辅助膜蛋白STA的计算。除了常规的膜法向量,用户还可以方便地通过MPicker在展平tomogram中标注蛋白在膜表面的面内取向,从而利用更强的角度约束来辅助STA的计算。为提高取向确定的自动化程度,研究团队将膜蛋白沿膜法向量投影,并进行了改造的二维分类,从而更高效地获得每个颗粒的精准取向信息。通过这一方法,团队使用四套tomogram数据成功解析了衣藻PSII-LHCII超复合物的低分辨原位结构(图3),并将结果上传至EMDB数据库(EMD-61019)。

图3.通过膜蛋白的法向量和面内角约束STA的计算
生命学院李雪明副教授为论文的通讯作者,生命学院2020级博士生颜啸峰为论文的第一作者。电子系李树东、黄炜琳,生命学院王皓博士(已毕业),电子系赵天放、黄铭涛,冷冻电镜平台周昵昀博士,以及电子系沈渊教授为本研究作出了重要贡献。研究得到国家自然科学基金委、生命科学联合中心、北京生物结构前沿研究中心等的资金支持,并得到国家蛋白质科学研究(北京)设施清华基地的技术支持。
论文链接:
https://doi.org/10.1038/s41467-024-55767-w
软件链接:
https://thuem.net/software/mpicker/overview.html
供稿:生命学院
编辑:李华山
审核:郭玲
